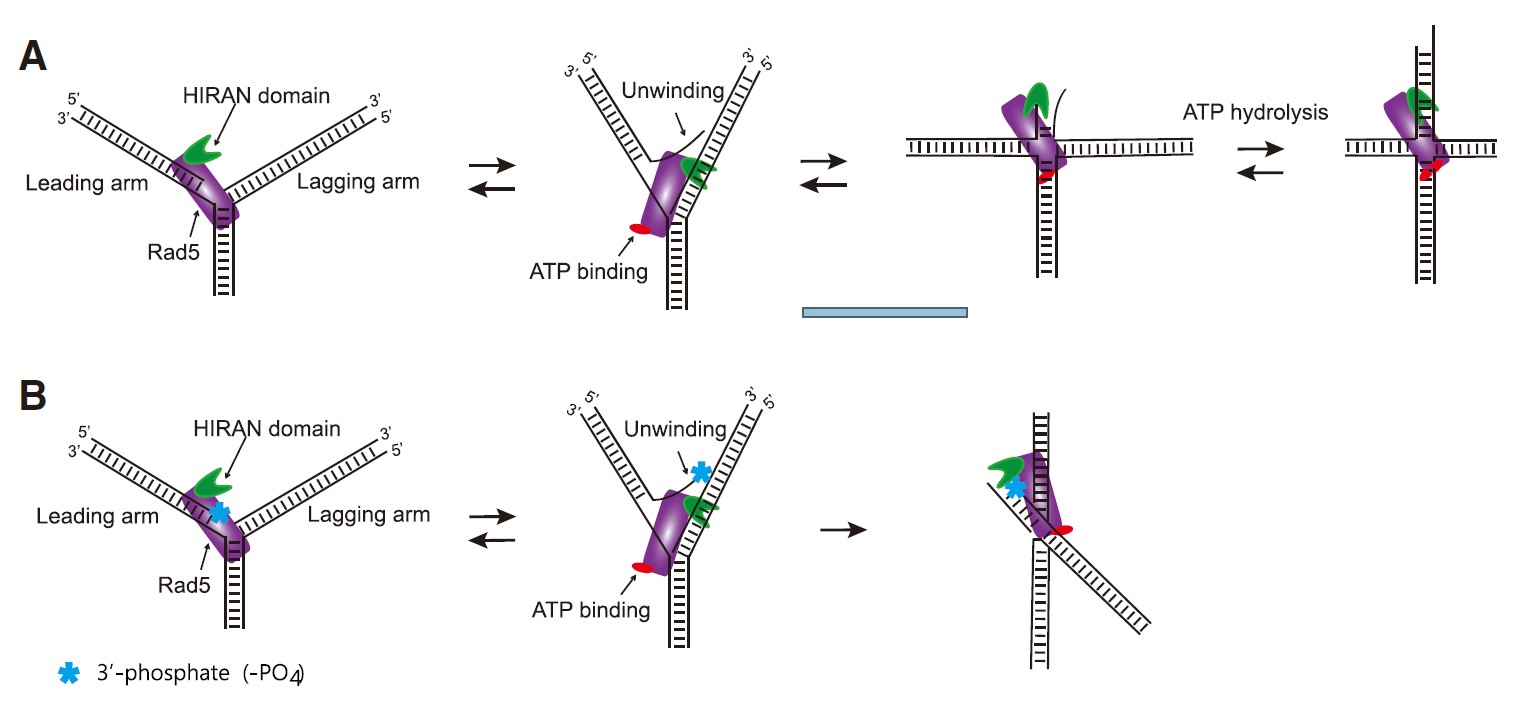
<Rad5의 DNA replication fork reversal 모델>
1. 배경지식
다양한 DNA lesion은 replication fork를 stall할 수 있습니다. 이러한 stalling은 genome의 instability 와 cell death를 일으킬 수 있기 때문에, stalled replication forks를 reactivation하는 기작을 세포는 가지게 됩니다. Replication fork reversal은 이러한 pathway들 중 핵심 초기 단계로 알려져 있습니다. Remodeling 과정 중 replication fork는, three-way junction에서 four-way junction으로 변하게 되고, 이후 DNA repair substrate로 사용됩니다. 그리고 DNA replication 재 시작을 위해, Revered 된 replication fork는 다시 tree-way junction으로 restored 됩니다.
박테리아 그리고 인간에 걸쳐 몇 효소들은 replication fork reversal activity를 가지고 있습니다. 이러한 motor protein들은 RecQ helicases, FANCM, 그리고 SWI/SNF helicases를 포함합니다 (Helicase superfamily 2). Replication fork를 four-way junction으로 remodeling하기 위해서는 leading 과 lagging arm들을 unwinding 하고, parental과 daughter strand를 annealing하는 2가지 다른 과정이 필요합니다.
2. 질문
Replication fork reversal activity를 갖는 효소들은 DNA double-helix unwinding activity를 당연히 갖고 있다 생각하지만, SWI/SNF motor 단백질인 Rad5가 별도의 DNA-unwinding activity를 갖고 있는지는 아직 밝혀지지 않았습니다. 따라서, SWI/SNF motor 단백질인 Rad5가 어떻게 remodeling process를 initiate할 수 있으며, DNA-unwinding activity를 갖고 있는가 알고 싶었습니다.
3. 발견
본 논문에서는Saccharomyces cerevisiae에서 유래한 Rad5의 replication fork reversal initiation 기작을 이해하기 위해, single-molecule FRET (fluorescence resonance energy transfer) 기술을 사용하였습니다. 순수 정제한 재조합 단백질과 model DNA replication forks를 사용하여 실험을 진행 하였습니다. 저희는 Rad5가 ATP에 binding 함에 따라 leading arm을 unwinding 하고 leading arm 과 lagging arm을 proximally positioning하는 것을 관찰하였습니다. Rad5는 four-way junction이 형성되면, ATP hydrolysis 에너지를 사용하여, 7.1 ± 0.14 nt/s 속도로 migration하는 것을 관찰 하였습니다. Rad5의 HIRAN domain이 leading arm의 3’ end 에 anchoring 되면, branch migration과 three-way junction recovery에 중요하지만, four-way junction에 구조적인 변화에는 영향이 없다는 것을 알 수 있었습니다.
4. 후속
다른 SWI/SNF helicases 단백질들이 Rad5와 같이 replication fork reversal mechanism이 conserve 되어 있는지 알아보면 좋을 것 같습니다.
5. 소감
생물 실험에 사용되는 대부분의 재료들은 단백질, DNA등 맨 눈으로 쉽게 볼 수 없는 작은 물질들 입니다. 따라서 실험을 함에 있어 각 물질들의 특성을 찾아 이해하고 실험의 과정들을 머릿속에서 상상으로 그려보며 실험을 하게 됩니다. 하지만, 이 논문에서는Single-molecule FRET 기술을 사용하여 실험에 사용되는 효소 및 substrate의 활성을 시간에 따라 볼 수 있다는 사실이 정말 좋았던 것 같습니다. 특히나, 문헌으로 찾은 ATPase 혹은 HIRAN domain mutant Rad5의 활성이, 논문에서 주장하고 싶은 방향으로 결과가 나온 것이 정말 기분이 좋았습니다.
6. 기타
새로운 분야를 공부 할 수 있게 기회를 주신 김재훈 교수님, 좋은 연구에 참여시켜주신 홍성철 교수님, 신수철 형에게 감사합니다.






 손유경(생체분자공학 연구실, 김학성 교수님)
손유경(생체분자공학 연구실, 김학성 교수님)
 김종원(생체분자공학 연구실, 김학성 교수님)
김종원(생체분자공학 연구실, 김학성 교수님)



















